纳米孔测序技术
纳米孔测序技术(Oxford Nanopore Technologies, 又称第四代测序技术)是最近几年兴起的新一代测序技术。
目前市场上广泛接受的纳米孔测序平台是Oxford Nanopore Technologies(ONT)公司的MinION,GridION X5 和 PromethION 三款不同类型测序仪。ONT测序的特点是单分子测序,测序读长长,测序速度快,测序数据实时监控,机器方便携带等。
据百迈客公布的不同物种实测数据显示:
基因组: 目前测序平均reads长度在20Kb以上,最长reads的N50高达48kKb左右,而最长reads可达到惊人的1.29M;
转录组: raw data数据量在2.5~4.4Gb,reads数为2.8~3.9M,其中N50在1.5kb左右,平均长度为1.0kb,平均质量值Q7以上。在对其进行质控后,其基因组比对率在85%左右。
读长和数据正确性是人们最为关心的两个方面,记得Nanopore测序器公布之初最大的缺陷就是超高的测序错误率 (5-15%),同样据百迈客公布结果显示经过ONT公司科研人员的不断努力,目前的测序结果不仅有超长的测序读长,下机数据碱基的平均准确率达到~86% (未经过纠错与polish的数据), 且与PacBio的准确率 (~85%)相当。而且组装后经过纠错与polish后碱基的准确度均在99.99%以上。
工作原理
在充满电解液的腔内,带有纳米级小孔的绝缘防渗膜将腔体分成2个小室,如图1,当电压作用于电解液室,离子或其他小分子物质可穿过小孔,形成稳定的可检测的离子电流。掌握纳米孔的尺寸和表面特性、施加的电压及溶液条件,可检测不同类型的生物分子。
由于组成DNA的四种碱基腺嘌呤(A)、鸟嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T)的分子结构及体积大小均不同,单链DNA(ssDNA)在核酸外切酶的作用下被迅速逐一切割成脱氧核糖核苷酸分子,当单个碱基在电场驱使下通过纳米级的小孔时,不同碱基的化学性质差异导致穿越纳米孔时引起的电流的变化幅度不同,从而得到所测DNA的序列信息。
测序过程
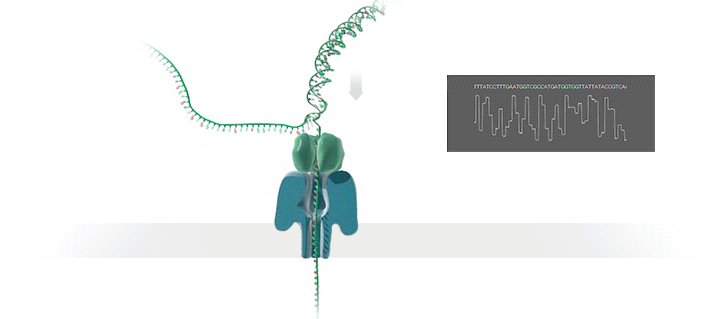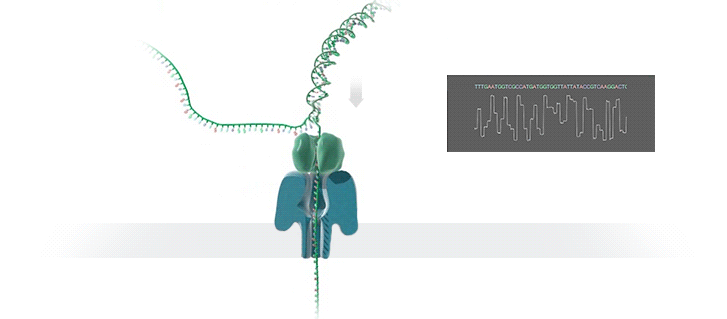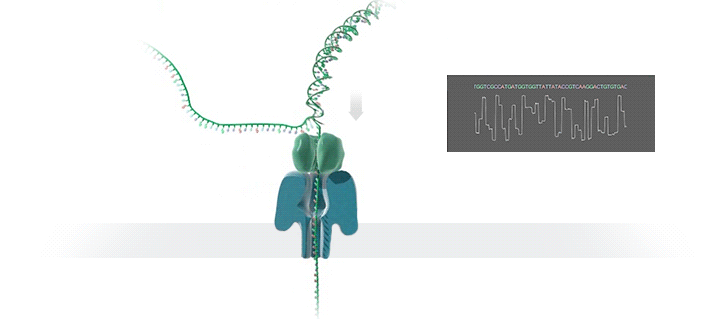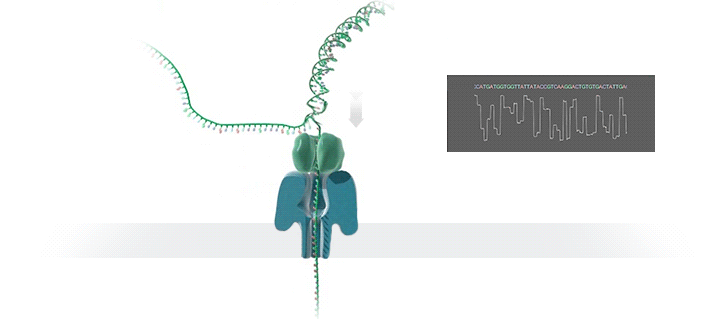
MinION纳米孔测序仪的核心是一个有2,048个纳米孔,分成512组,由专用集成电路控制的flow cell。测序原理见下图a所示:首先,将双分子DNA连接lead adaptor(蓝色),hairpin adaptor(红色)和trailing adaptor(棕色);当测序开始,lead adaptor带领测序分子进入由酶控制的纳米孔,lead adaptor后是template read(即待测序的DNA分子)通过纳米孔,hairpin adaptor的作用是DNA双链测序的保证,然后complement read(待测序分子的互补链)通过纳米孔,最后是trailing adaptor通过。在上述测序方法中,template read和complement read依次通过纳米孔,利用pairwise alignment,它们组合成2D read;而在另外一种测序方法中,不使用hairpin adaptor,只测序template read,最终形成1D read。后一种测序方法通量更高,但是测序准确性低于2D read。每个接头序列(adaptor)通过纳米孔引起的电流变化不同(图1c),这种差别可以用来做碱基识别。
测序平台简介
MinION
ONT最知名产品,仅有U盘大小,插入普通PC电脑即可运行,曾登上国际空间站,完成太空测序;MinION一次可运行一个flow cells,产出约10-30Gb的测序数据,运行时长约48小时;
GridION
GridION X5系统的测序部分包含五个 flow cells,这些flow cells可单独使用或协同使用,并通过USB连接到计算机。利用现有的试剂和软件每运行48小时可生成高达150GB的测序数据。GridION X5的出现填补了MinION和PromethION之间的空白。
PromethION
PromethION是ONT推出的最新款超高通量测序设备,它支持实时、长读长、直接DNA和RNA测序工作流程。PromethION一次可运行24 (PromethION 24) 到48 (PromethION 48) 个测序芯片,按照每张测序芯片包含3,000个纳米孔通道,所有芯片同时运行将可产出高达7.6Tb甚至是15Tb的数据,这能够满足超高通量的、快速周转测序需求。适用于大规模群体遗传学研究和大型动植物基因组测序项目;
应用领域
基因组组装
【Nanopore sequencing The advantages of long reads for genome assembly】
| Assembler name | Algorithms | Errorcorrection | Link | Reference |
|---|---|---|---|---|
| LQS | DALIGNER, Celera OLC | Nanocorrect, Nanopolish | https://github.com/jts/nanopolish | Loman (2015) |
| Canu | MHAP, Celera OLC | Canu | https://github.com/marbl/canu | Berlin (2015) |
| Canu | MHAP, Celera OLC | Racon, Pilon | https://github.com/nanoporetech/ont-assembly-polish | nanoporetech |
| Miniasm | OLC | https://github.com/lh3/minia | Li (2016) | |
| Miniasm | OLC | Racon | https://github.com/isovic/racon | Vaser (2017) |
| Ra-integrate | OLC | https://github.com/mariokostelac/ra-integrate/ | Sovic (2016) | |
| ALLPATHS-LG | de Bruijn graph | ALLPATHS-LG | https://www.broadinstitute.org/software/allpathslg/blog/?page_id | Gnerrea (2011) |
| SPAdes | de Bruijn graph | SPAdes | http://bioinf.spbau.ru/spades | Bankevich (2012) |
| SMART denovo | Smith-Waterm, dot matrix | https://github.com/ruanjue/smartdenovo | Ruan | |
| ABruijn | de Bruijn graph | https://github.com/fenderglass/ABruijn | Lin (2016) |
宏基因租
【Nanopore sequencing Addressing the challenges of metagenomics for environmental and clinical research】
变异分析
【Nanopore sequencing The application and advantages of long-read nanopore sequencing to structural variation analysis】
甲基化分析
结合ONT单分子长读长的实时测序特性,ONT甲基化数据能够真实的还原碱基修饰信息,得到准确和类型丰富的检测结果;
参考资料
- 纳米孔测序技术发展简介
- 全面解读第四代测序技术
- Jain, Miten, et al. “The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community.“ Genome biology 17.1 (2016): 239.
- Leggett, Richard M., and Matthew D. Clark. “A world of opportunities with nanopore sequencing.” Journal of Experimental Botany (2017): erx289.
