之前我们也了解过Single Cell 全基因组扩增过程,最近北大的谢老师又重新刷新了单细胞全基因组扩增的新高度:Single-cell whole-genome analyses by Linear Amplification via Transposon Insertion (LIANTI),下面对本文做简单的解读?
转座子插入的线性扩增
Linear Amplification via Transposon Insertion (LIANTI)
Combines Tn5 transposition and T7 in vitro transcription for single-cell genomic analyses;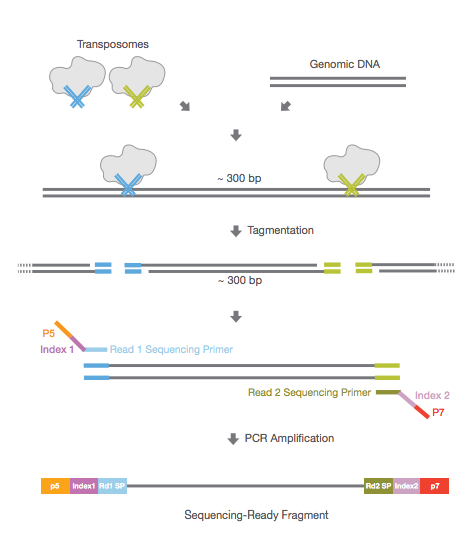
指数扩增 Vs 线性扩增
线性扩增优于指数扩增,基于以下两方面考虑: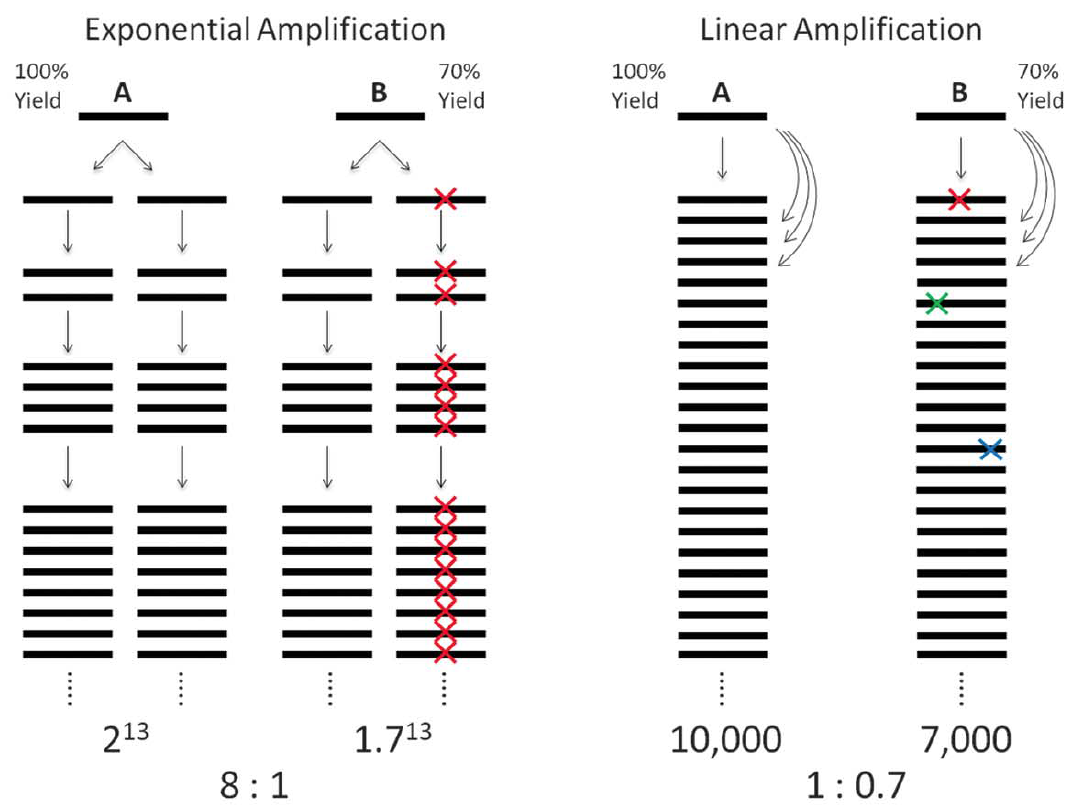
拷贝数
例如,上图中假设DNA片段A和B的扩增效率 (replication yields) 分别为100%和70%每一次,并假设原始A/B=1:1,最终的扩增系数 (amplification factor) 为片段A 大约10,000;
即在指数扩增 (上一次的扩增结果可以成为下一次扩增的模版) 时需要经过13次扩增过程 (213=8,192; 214=16,384),此时B对应的最终产物量为1.713~=990,A/B~=8:1;
而线性扩增仅使用最初模版,扩增出的模版被分离出来不进入扩增过程,所以需要10,000次的扩增过程,此时B对应的最终产物量为0.710,000=7,000,A/B=1:0.7;
*当上述扩增用于研究拷贝数变异(CNV)时,指数扩增会引起致命性错误;
准确性fidelity
对于保真性达10-7的高保真聚合酶扩增一次人的基因组 (3X 109bp)理论上将随机性的引入大约300个碱基的错误,并且因为指数扩增特性这300个碱基错误的扩增产物将作为下一次扩增的模版,在300个碱基错误的基础上可能再次引入300个碱基的错误,而且上一次扩增的错误会延续在下一次扩增中,这样循环下去这种错误将会被无限次的扩大,这在检测SNVs时会产生假阳性。
相反,在线性扩增时,由于模版始终为最初的模版,所以这种扩增错误每次都会随机出现在不同的位置,很容易通过不同时期扩增产物间的比对而消除。
SNP Vs SNV
单核苷酸多态性(single nucleotide polymorphism,SNP) 和单核苷酸位点变异(single nucleotide variants, SNV)。个体间基因组DNA序列同一位置单个核苷酸变异(替代、插入或缺失)所引起的多态性。不同物种、个体基因组DNA序列同一位置上的单个核苷酸存在差别的现象。有这种差别的基因座、DNA序列等可作为基因组作图的标志。人基因组上平均约每1000个核苷酸即可能出现1个单核苷酸多态性的变化,其中有些单核苷酸多态性可能与疾病有关,但可能大多数与疾病无关。单核苷酸多态性是研究人类家族和动植物品系遗传变异的重要依据。在研究癌症基因组变异时,相对于正常组织,癌症中特异的单核苷酸变异是一种体细胞突变(somatic mutation),称做SNV。
SNP (single nucleotide polymorphism) vs. SNV (single nucleotide variant) As their name suggests, both are concerned with aberrations at a single nucleotide. However, a SNP is when an aberration is expected at the position for any member in the species 鈥?for example, a well characterized allele. A SNV on the other hand is when there is a variation at a position that hasn鈥檛 been well characterized 鈥?for example, when it is only seen in one individual. It is really all a question of frequency of occurrence.
扩增过程原理
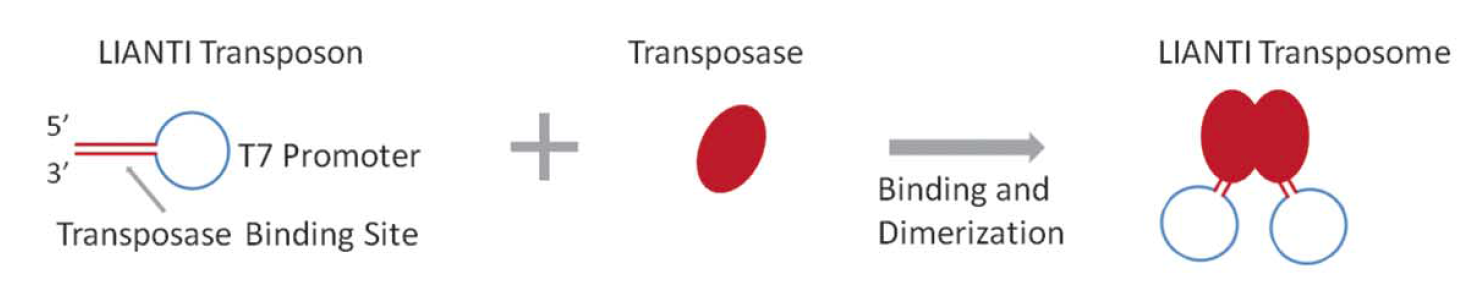
1.在LIANTI扩增时来自于单细胞的基因组DNA在Tn5转座酶的作用下转座LIANTI转座子 而随机片段化(400bp左右)。
- LIANTI转座子 (LIANTI transposon):包含一段19bp双链的转座酶结合位点和单链的T7启动子环;
- LIANTI transposon DNA (5’/Phos/CTGTCTCTTATACACATCTGAACAGAATTTAATACGACTCACTATAGGGAGATGTGTATAAGAGACAG-3’, IDT oligo with PAGE purification) ;
- 等摩尔量的LIANTI 转座子和Tn5转座酶 (Tn5 transposase) 混合形成二聚体的LIANTI转座体 (LIANTI transposome).
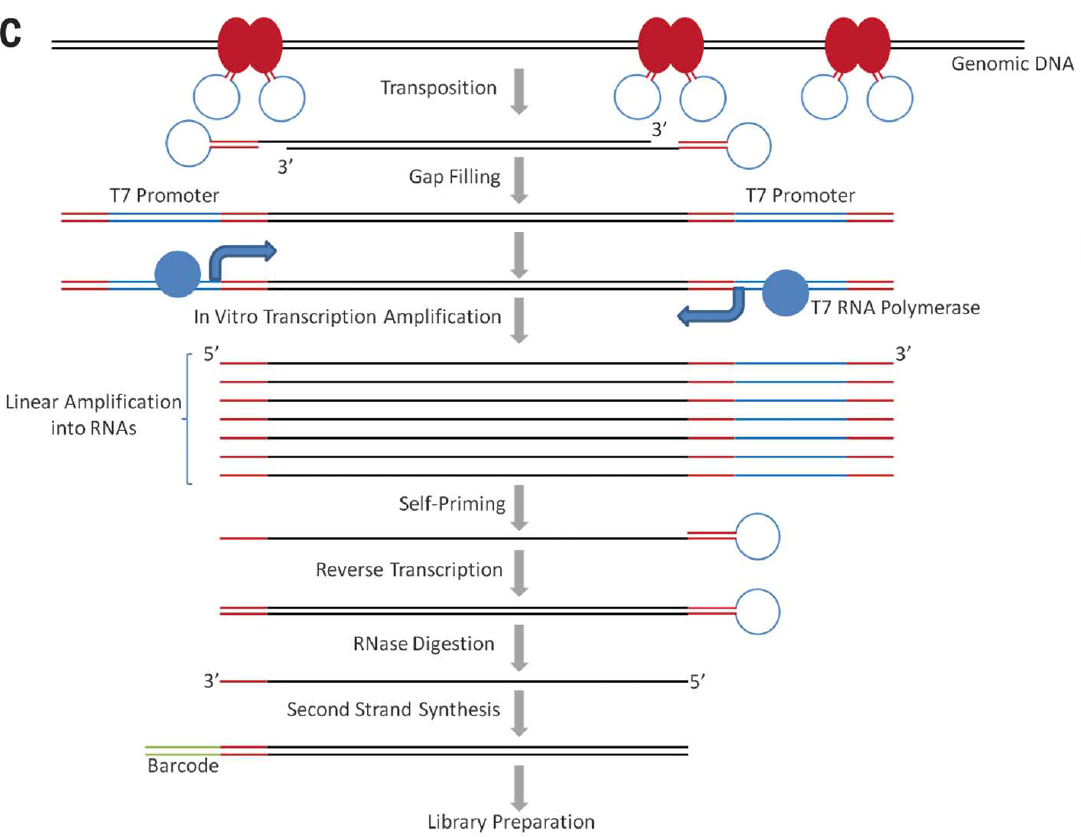
2.碎片化后的基因组DNA被加上T7启动子标签,随后在体外转录线性扩增成成千上万的RNAs,紧接着3’端反向 and 合成互补的第二链形成双链LIANTI扩增用于DNA文库。
- 来源于单细胞的基因组DNA被随机碎片化后被LIANTI转座子标记,随后DNA聚合酶作用于碎片DNA双链两端互补单链的环状T7启动子为双链的T7启动子;
- 在T7RNA聚合酶的作用下体外转录线性扩增基因组DNA为基因组RNA,其中转录出的RNA能够在3‘ 端自动环化使单链状转座酶结合位点形成双链状;
- 随后经历反转录作用,RNase消化和第二链的合成,双链的LIANTI扩增物标记上特异的分子条形码 (unique molecular barcodes) 代表原始单细胞基因组DNA 的扩增产物用于之后的DNA文库和高通量测序;
LIANTI扩增消除了非特异性priming和指数扩增相较于其他全基因组扩增方法,因此能够大大的降低扩增的偏好性和错误性。
相关疑问
1. 碎片化真的随机吗?
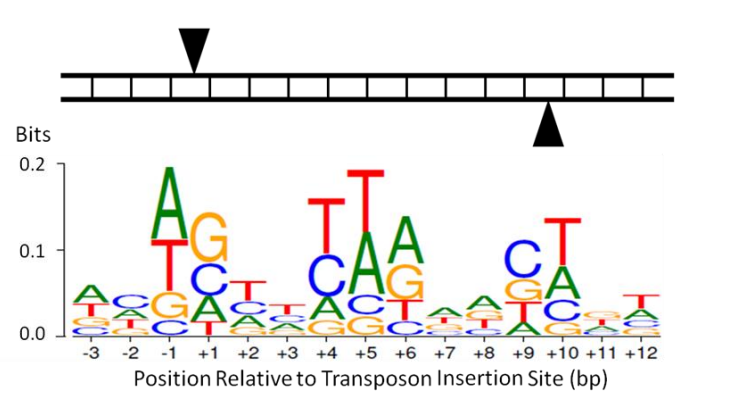
上图中黑色三角表示单细胞基因组插入碎片化位点,WebLogo表明其插入位点碱基不存在明显的保守性。
WebLogo说明:以下序列对应WebLogo图如下👇
2
3
4
5
6
7
attcgtgatagctgtcgtaaag
>2
ttttgttacctgcctctaactt
>3
tgccgtgattatagacactttty axis解释the height of the y-axis is the maximum entropy for the given sequence type. (log2 4 = 2 bits for DNA/RNA, log2 20 = 4.3 bits for protein.)【WebLogo: A Sequence Logo Generator】
2. 碎片DNA大小?
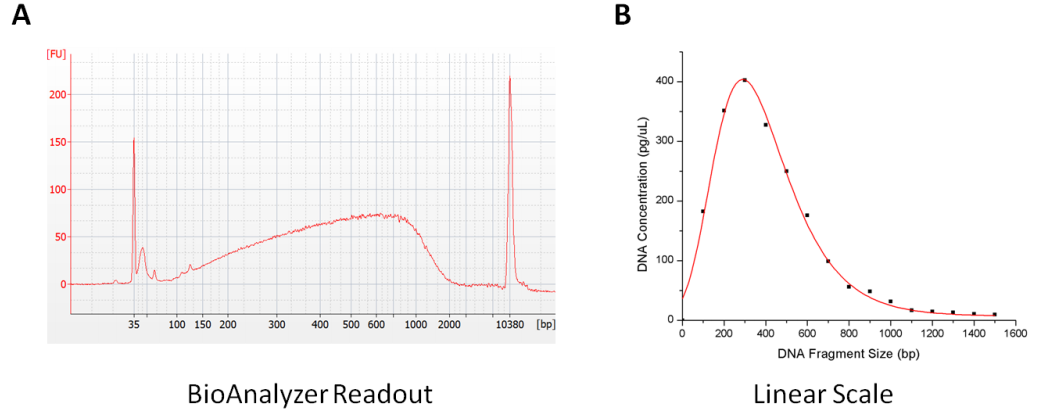
上图表明单细胞基因组被LIANTI转座体随机碎片化后平均碎片大小约为400bp。
3. 扩增效率?
对于单个包含~ 6 pg基因组DNA的人类细胞,体外转录扩增过夜可得到~20 ng的LIANTI扩增DNA,随后进行~30测序,并与其他扩增方法比较,发现:LIANTI扩增达到97%的全基因组覆盖度和17%的allele dropout rate。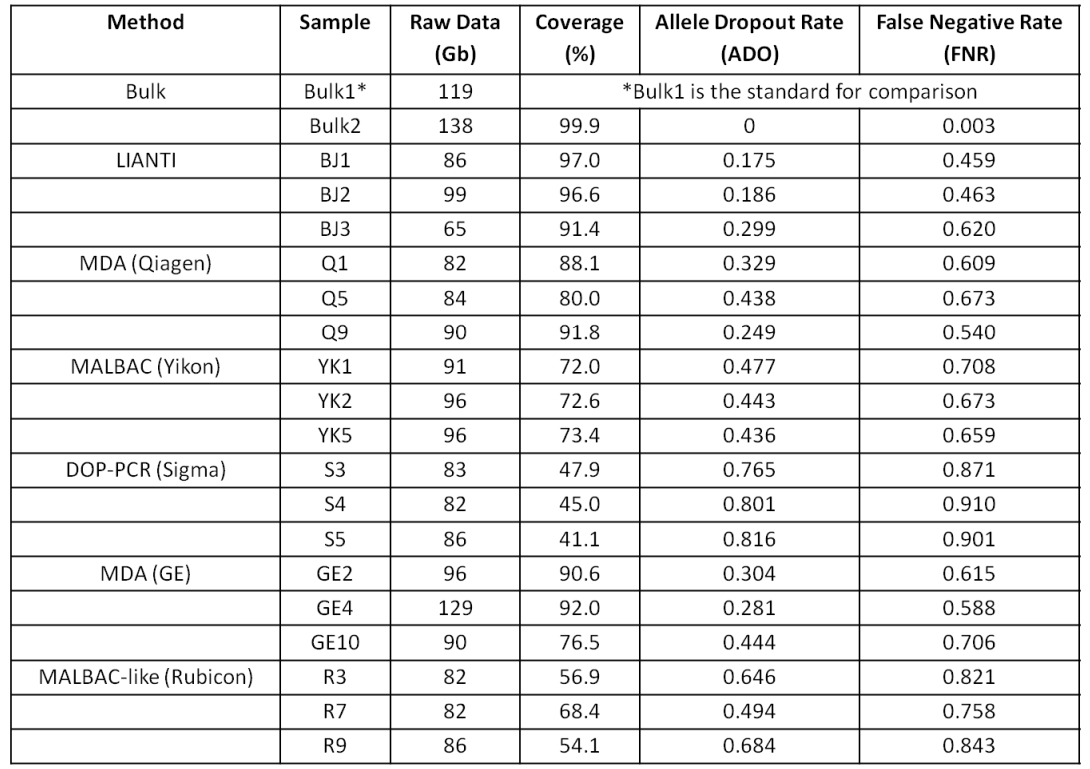
4. 扩增的一致性 (amplification uniformity) 如何?
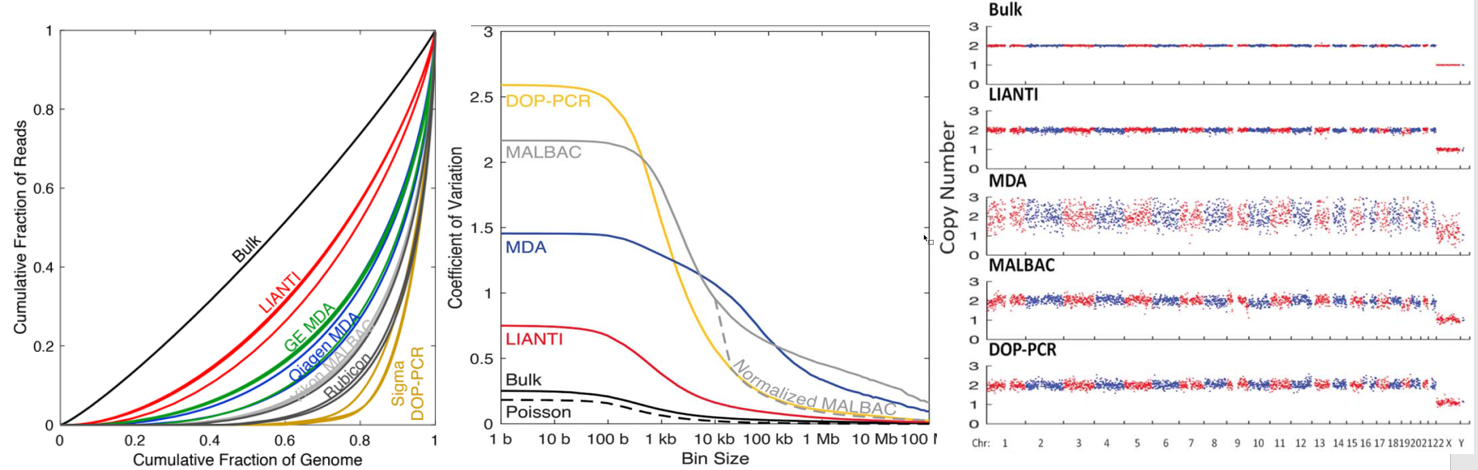
上图表明LIANTI表现出较高的扩增一致性和较低的变异系数。
Lorenz curve:Perfectly uniform coverage leads to the diagonal line, and deviation from the diagonal line represents amplification bias.
变异系数coefficient of variation (CV):标准差与平均数的比值,无量纲,用来反映数据离散程度的绝对值。
相关参考文献
- Simul-seq: combined DNA and RNA sequencing for whole-genome and transcriptome profiling
- Scalable whole-genome single-cell library preparation without preamplification