Illumina二代测序有个致命缺陷,说到底还是基于PCR扩增的,所以存在偏向性和对于高GC含量区无法扩增等系统误差,测序错误是不可避免的,其次就是测序长度短;但其价格便宜,通量非常高,准确性达99%,综合性价比也受到青睐。短序列的reads在做基因组装的时候,遇到大的重复片段就会很吃力。
10X Genomics
2015年备受瞩目的测序黑马:10X Genomics,是常规Illumina二代测序的升级版,由于开发出了一套巧妙的Barcoding建库方案,使得Illumina这种短读长二代测序能够得到跨度在30-100Kb的linked reads信息,与二代测序数据相结合,在Scaffold的组装上能够得到媲美三代测序的组装结果;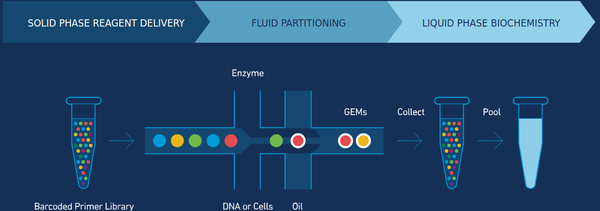
基本原理: 首先将每一条长片段的DNA分配至不同的油滴微粒中,通过专利的GEM建库技术,长片段DNA被切碎成适合测序的大小,并且来源于相同油滴(同一条长片段DNA)的DNA片段,会带上相同的一段DNA序列标记(Barcode),之后在Illumina系统上测序完成后,可以理论上再将来源相同的DNA序列独立拼接,得到原先的长片段DNA序列。
对于不同GC含量区其效果如何呢?2015年10月Nat Review Genetics文章Genetic variation and the de novo assembly of human genomes中总结的PacBio、10X Genomics以及Illumina技术在不同GC含量DNA区域的覆盖度分布: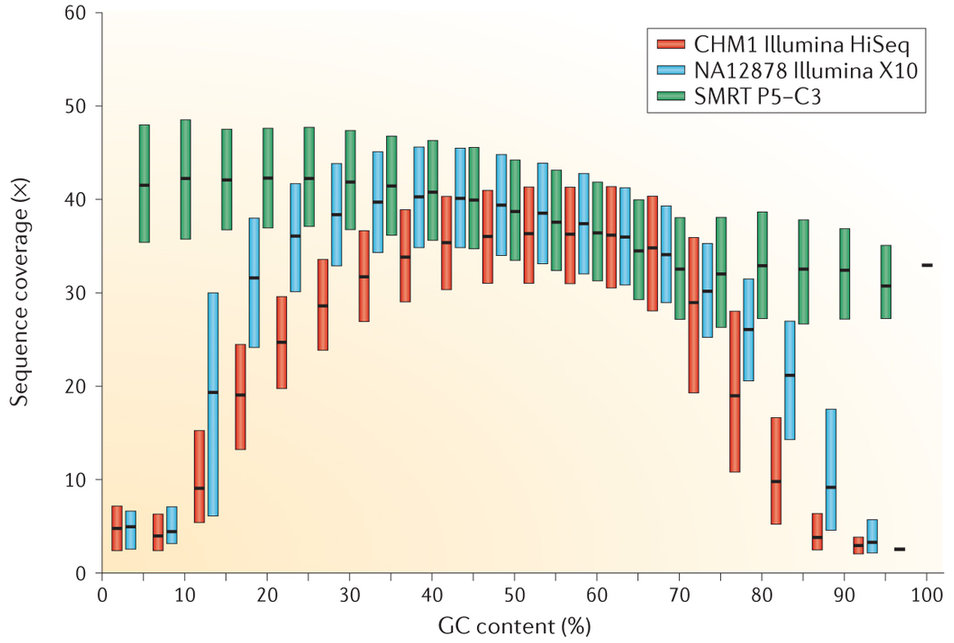
10X Genomics技术相对于Illumina来说,有改进,但依旧是个拱形,而PacBio则是无偏倚的均一分布,10X的技术,其Coverage一样是受GC含量影响较大的,那么如果真要应用10X技术,那么必须注意目标DNA的GC含量分布最好能控制在30~70%。
但10Xgenome毕竟是升级版,其也存在一些特有的优势:
PacBio
PBcR: 混合纠错拼接
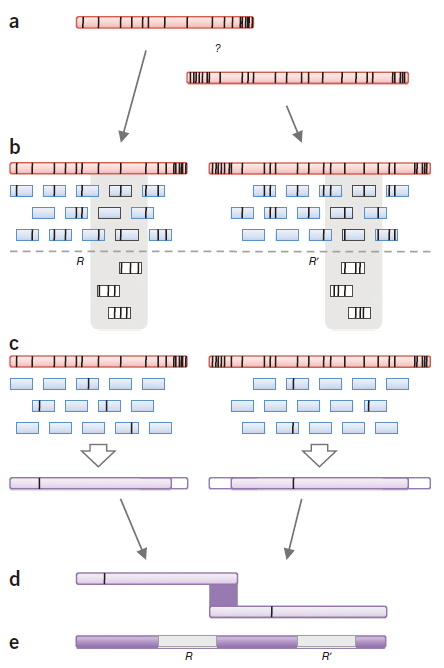
PBcR: 混合纠错拼接 粉色长方形:单个PacBio RS reads;黑色竖线:测序错误;(a)由于测序错误碱基的存在使得两条reads就难确定是否在末端重叠;(b)高质量的短reads比对到存在错误的长reads;短reads中的黑色竖线表示 ‘mapping errors’ ,是长reads和短reads中测序错误的组合,此外双拷贝的重复序列的存在(灰色轮廓)导致在每一个拷贝中出现短reads的堆挤,为避免reads map到错误的重复区,仅保留最高比对值的短reads;(c)剩余的比对形成一致性序列(紫色长方形),长reads和短reads中共有的部分错误未能得到纠正;(d)overlap纠正后的长reads;(e) 最后的组装能够跨越重复区域。
Illumina reads纠错覆盖度
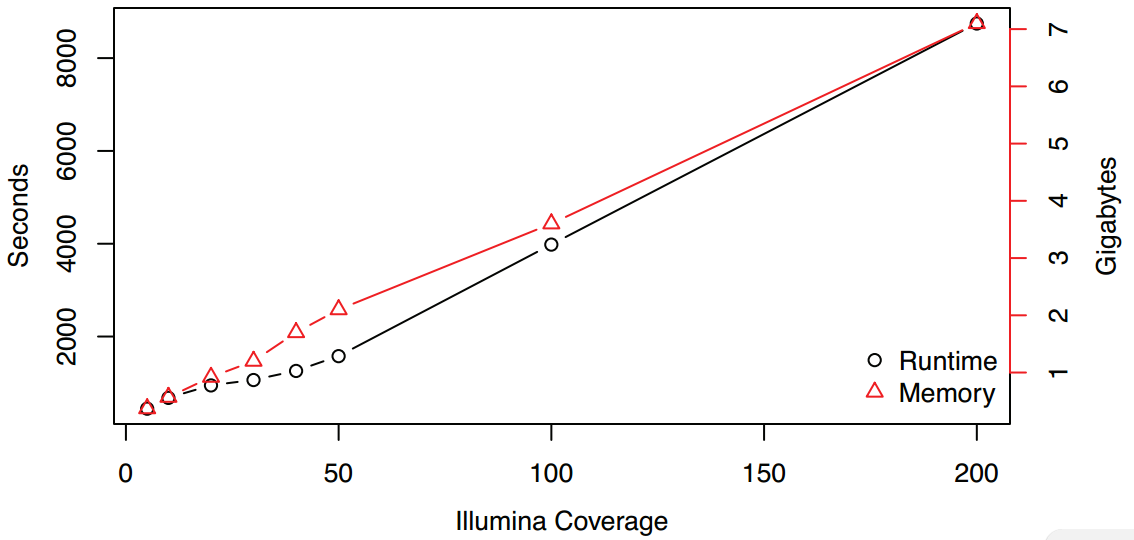
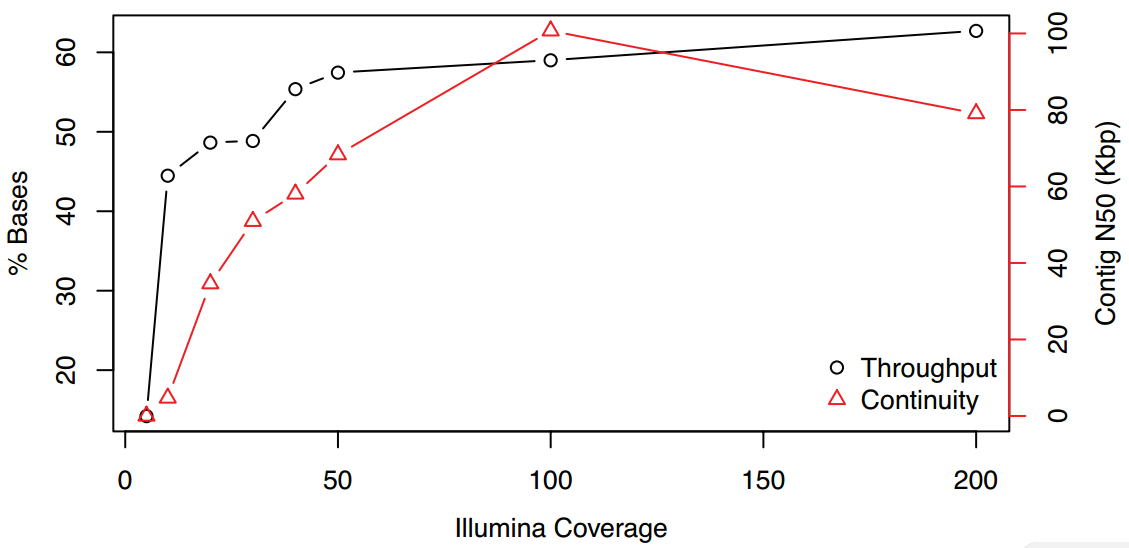
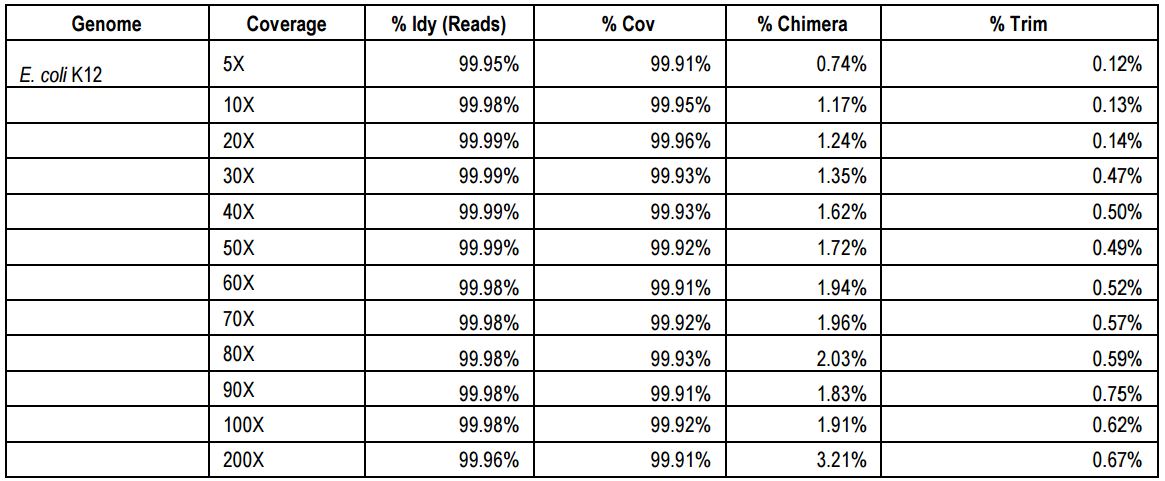
纠错的准确性和组装一致性在Illumina高质量reads达50X后开始收益递减,因此50X Illumina reads足够,纠错后PacBio长reads准确性将由85%提升至>99.9%,此时嵌合体和错误剪切reads分别为<2.5% 和 <1%。
目前在P6C4试剂下,大约每SMRT Cell平均可以做到 600M~1G数据量。
PacBio的长读长、无GC偏向性和无PCR扩增偏向性等独特优势有助于克服复杂的重复区域,从而跨越整个基因转录区,显著提升基因组和转录组的De Nove组装质量;
Illumina二代+PacBio三代数据分析
PBcR首先通过纠错来提升PacBio reads准确性,然后进行组装。PBcR的纠错和组装分为self-correction (using only PacBio RS data,自动运行fastqToCA) or correction with high-identity sequences(二代数据)。
self-correction
1 | PBcR -length 500 -partitions 200 -l lambda -s pacbio.spec -fastq pacbio.filtered_subreads.fastq genomeSize=50000 > run.out 2>&1 |
高质量Illumina reads
1 | #short read准备 |
: 第一步short reads准备阶段请确认二代数据第四行质量编码值,一般是33,否则用-type参数指定,要不然会报错QV问题;
纠正时PBcR需要安装AMOS和blasr依赖软件,输入文件short reads (illumina.frg)和long reads (pacbio.filtered_subreads.fastq);
fastqToCA和PBcR两个中的libraryname需不同;
fastqToCA生成的frg文件后面没有序列信息 ,是正确的;
Spec files参数解释
PBcR混合组装需要指定两个Spec配置文件: pacbio.spec(纠错)和asm.spec(组装)。这两个文件都包含特定的算法参数和计算机硬件参数,通常情况下算法参数可以忽略(此时将用软件默认值),但是计算机硬件参数需要根据实际情况调整。
所有参数均为option = value形式,其中的value为布尔型(boolean),即true=1,false=0。
具体关于specfile参数解释见PBcR:SpecFiles Options
Spec files实例
集群下参考pacbio.spec
1 | #以下为grid计算 |
集群下参考asm.spec
1 | ######################以下为gird计算,useGrid = 1 #################### |
输出结果
9-terminator;参考资料
当10X Genomics遇上PacBio——烫金开始剥落了
第三代测序成本偏高是什么原因导致的?
Computational Science Community Wiki: Sun Grid Engine: Job Arrays
