
纳米孔测序技术
纳米孔测序技术(又称第四代测序技术)是最近几年兴起的新一代测序技术。目前测序长度可以达到150kb;
目前市场上广泛接受的纳米孔测序平台是Oxford Nanopore Technologies(ONT)公司的MinION纳米孔测序仪。它的特点是单分子测序,测序读长长(超过150kb),测序速度快,测序数据实时监控,机器方便携带等。
纳米孔分析技术起源于Coulter计数器的发明以及单通道电流的记录技术。生理与医学诺贝尔奖获得者Neher和Sakamann在1976年利用膜片钳技术测量膜电势,研究膜蛋白及离子通道,推动了纳米孔测序技术的实际应用进程。1996年,Kasianowicz 等提出了利用α-溶血素对DNA测序的新设想,是生物纳米孔单分子测序的里程碑标志。随后,MspA孔蛋白、噬菌体Phi29连接器等生物纳米孔的研究报道,丰富了纳米孔分析技术的研究。Li等在2001年开启了固态纳米孔研究的新时代。经过十几年的发展,现如今固态纳米孔技术日益发展成熟。
目前用于DNA测序的纳米孔有两类:生物纳米孔(由某种蛋白质分子镶崁在磷脂膜上组成)和固态纳米孔(包括各种硅基材料、SiNx、碳纳米管、石墨烯、玻璃纳米管等)。DNA链的直径非常小(双链DNA直径约为2nm,单链DNA直径约为1nm),对所采用的纳米孔的尺寸要求较苛刻。
工作原理
在充满电解液的腔内,带有纳米级小孔的绝缘防渗膜将腔体分成2个小室,如图1,当电压作用于电解液室,离子或其他小分子物质可穿过小孔,形成稳定的可检测的离子电流。掌握纳米孔的尺寸和表面特性、施加的电压及溶液条件,可检测不同类型的生物分子。
由于组成DNA的四种碱基腺嘌呤(A)、鸟嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T)的分子结构及体积大小均不同,单链DNA(ssDNA)在核酸外切酶的作用下被迅速逐一切割成脱氧核糖核苷酸分子,当单个碱基在电场驱使下通过纳米级的小孔时,不同碱基的化学性质差异导致穿越纳米孔时引起的电流的变化幅度不同,从而得到所测DNA的序列信息。
测序过程
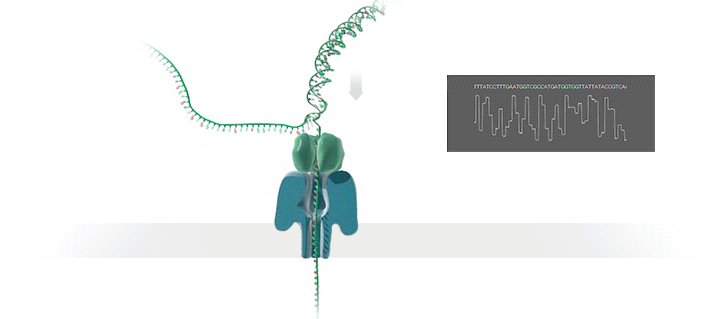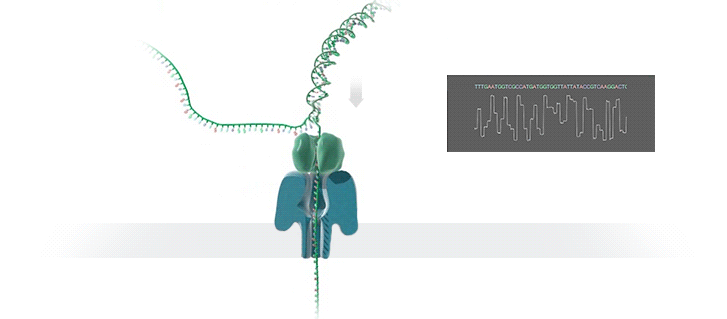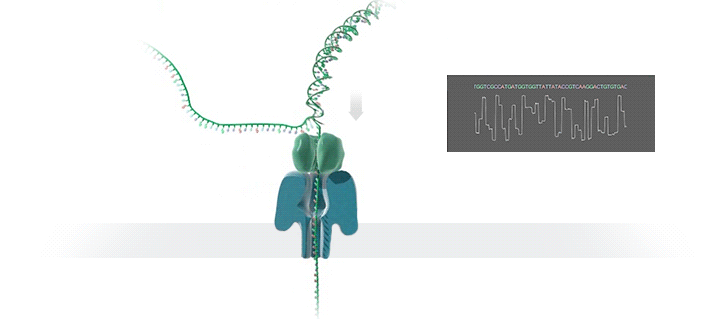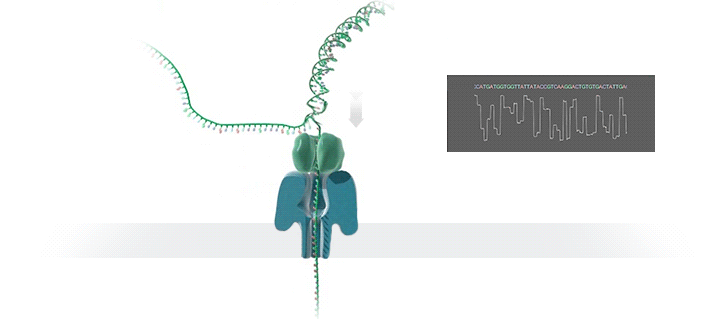
Nanopore sequencing: (a) A biological nanopore is inserted into an electrically resistant synthetic membrane. A potential is applied across the membrane, resulting in ion flow. Library DNA molecules have adaptors with aliphatic tethers (not shown) which preferentially locate to the membrane for a localized library concentration. (b) The motor protein bound to the other adaptor docks with the pore, and passes the DNA molecule through it. (c) Bases in the nanopore cause disruptions in the current which are characteristic of their sequence (blue line). In some basecallers, the signal is further refined to events (red line) which correspond to distinct pore kmers.
MinION纳米孔测序仪的核心是一个有2,048个纳米孔,分成512组,由专用集成电路控制的flow cell。测序原理见下图a所示:首先,将双分子DNA连接lead adaptor(蓝色),hairpin adaptor(红色)和trailing adaptor(棕色);当测序开始,lead adaptor带领测序分子进入由酶控制的纳米孔,lead adaptor后是template read(即待测序的DNA分子)通过纳米孔,hairpin adaptor的作用是DNA双链测序的保证,然后complement read(待测序分子的互补链)通过纳米孔,最后是trailing adaptor通过。在上述测序方法中,template read和complement read依次通过纳米孔,利用pairwise alignment,它们组合成2D read;而在另外一种测序方法中,不使用hairpin adaptor,只测序template read,最终形成1D read。后一种测序方法通量更高,但是测序准确性低于2D read。每个接头序列(adaptor)通过纳米孔引起的电流变化不同(图1c),这种差别可以用来做碱基识别。
分析工具

目前针对Nanopore测序数据的生物信息分析工具已经研发,包括最新的国产三代数据组装软件 MECAT和老牌的 MaSuRCA等,详细支持软件目录见:https://nanoporetech.com/resource-centre/tools。
主要应用领域
1. 基因组组装
【Nanopore sequencing The advantages of long reads for genome assembly】
| Assembler name | Algorithms | Errorcorrection | Link | Reference |
|---|---|---|---|---|
| LQS | DALIGNER, Celera OLC | Nanocorrect, Nanopolish | https://github.com/jts/nanopolish | Loman (2015) |
| Canu | MHAP, Celera OLC | Canu | https://github.com/marbl/canu | Berlin (2015) |
| Canu | MHAP, Celera OLC | Racon, Pilon | https://github.com/nanoporetech/ont-assembly-polish | nanoporetech |
| Miniasm | OLC | https://github.com/lh3/minia | Li (2016) | |
| Miniasm | OLC | Racon | https://github.com/isovic/racon | Vaser (2017) |
| Ra-integrate | OLC | https://github.com/mariokostelac/ra-integrate/ | Sovic (2016) | |
| ALLPATHS-LG | de Bruijn graph | ALLPATHS-LG | https://www.broadinstitute.org/software/allpathslg/blog/?page_id | Gnerrea (2011) |
| SPAdes | de Bruijn graph | SPAdes | http://bioinf.spbau.ru/spades | Bankevich (2012) |
| SMART denovo | Smith-Waterm, dot matrix | https://github.com/ruanjue/smartdenovo | Ruan | |
| ABruijn | de Bruijn graph | https://github.com/fenderglass/ABruijn | Lin (2016) |
2. 宏基因租
【Nanopore sequencing Addressing the challenges of metagenomics for environmental and clinical research】
3. 变异分析
【Nanopore sequencing The application and advantages of long-read nanopore sequencing to structural variation analysis】
PacBio vs. Oxford Nanopore sequencing

二者是目前主要的长reads测序技术,且具同样的缺点:高的错误率,但基于二者测序原理的差异,相较于Oxford Nanopore(MinION测序数据目前只有92%的准确性)而言PacBio错误率较低;就测序通量而言,Oxford Nanopore可同时测序多个分子,固通量较高;Oxford Nanopore测序仪体积仅有U盘大小,便于携带且测序费用低;
参考资料
- 纳米孔测序技术发展简介
- Jain, Miten, et al. “The Oxford Nanopore MinION: delivery of nanopore sequencing to the genomics community.“ Genome biology 17.1 (2016): 239.
- Leggett, Richard M., and Matthew D. Clark. “A world of opportunities with nanopore sequencing.” Journal of Experimental Botany (2017): erx289.
- PacBio vs. Oxford Nanopore sequencing
