使用场景
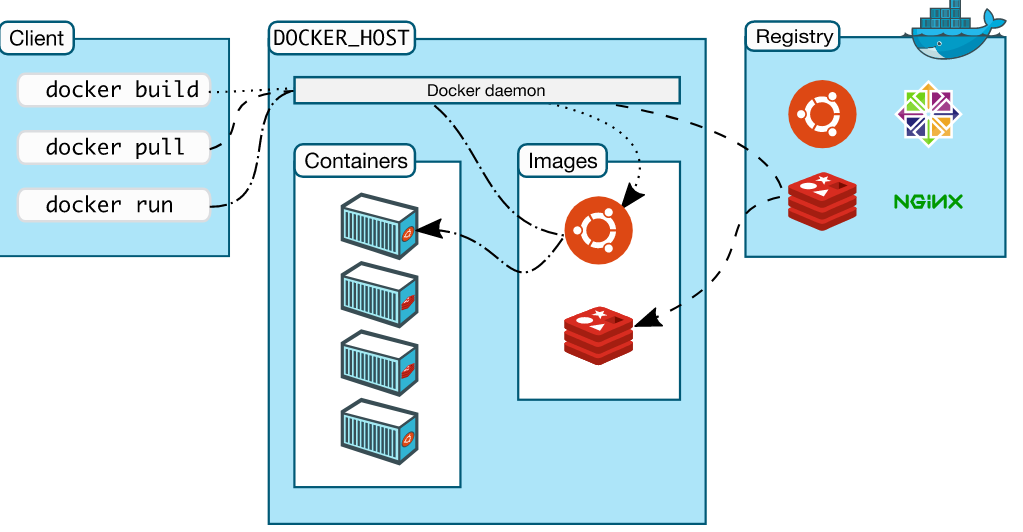
容器作为轻量级的虚拟机,可在主机之外提供多种系统环境选择;另外,在容器中一次打包好软件及相关依赖环境之后,即可将复杂的软件环境在各种平台上无缝运行,无需重复多次配置,大大减轻相关工作人员的工作量;
目前主流的容器为docker,其最初被用于软件产品需要快速迭代的互联网行业,极大地简化了系统部署、提高了硬件资源的利用率,近来也在各种特定领域的应用系统中被使用。
Docker 是一个开源的应用容器引擎,基于 Go 语言,并遵从Apache2.0协议开源。
Docker 可以让开发者打包他们的应用以及依赖包到一个轻量级、可移植的容器中,然后发布到任何流行的 Linux 机器上,也可以实现虚拟化。
容器是完全使用沙箱机制,相互之间不会有任何接口(类似 iPhone 的 app),更重要的是容器性能开销极低。
Docker 的优点:
- 比较轻量级:相对于虚拟机这样的技术来说,docker 比较轻量级,占用的额外资源少
- 容易维护: 采用Docker file 定义环境,方便更新和共享
- 操作简单: Docker 的操作非常的简单,学习成本低
在生物信息领域,为了一次配置,多次使用的目的,一些复杂的软件开始使用docker进行打包分发,另外由于云的兴起,docker也用于搭建私有生物信息云分析平台。尽管如此,由于权限、资源管理等因素限制,docker并未在传统HPC集群(物理裸机+操作系统+作业调度系统+高速互联网络)中流行开。
于是,有人开发了专门针对传统HPC集群的容器工具singularity,其使得普通用户可以方便地在集群使用打包好的容器镜像,配合作业调度系统,其使用也非常方便,跟使用其它应用软件的方式相同。singularity还有一大优势是,可直接使用docker镜像,大大提高了singularity的可用性。
安装
Docker和Singularity的服务器安装需要Root权限,所以可选择在本地个人电脑安装Docker用来制作镜像,然后上传服务器用Singularity运行;
镜像集合
Docker Hub
Singularity Hub
BioContainers,一个类似Bioconductor、Bioconda的生物信息分析工具集合。
Docker 国内镜像站
DaoCloud: 为了解决国内用户使用 Docker Hub 时遇到的稳定性及速度问题 DaoCloud 推出永久免费的新一代镜像站服务。
Docker 应用实例
镜像搜索
使用 docker search 命令来搜索标星至少为 25 的 CentOS 相关镜像。1
docker search -f stars=25 centos
下载一个镜像到本地1
2
3
4 下载远程镜像
docker pull biocontainers/bowtie2
载入本地镜像
docker load docker_rnaseq_ref.tar
察看所下载镜像1
docker images
察看docker运行和相关软件镜像对应参数1
2docker run --help
docker run biocontainers/bowtie2
其中重要参数有:
--rm:使得该容器退出后自动删除;-v:进行本地文件夹的挂载,用来指定输入数据位置和输出结果位置;
删除本地镜像1
2docker image rm [选项] <镜像1> [<镜像2> ...]
docker image rm biocontainers/bowtie2
[选项]的 -f 参数来执行强制删除;
Docker 创建自己的镜像
构建镜像的方式有好几种,我们主要利用Dockerfile,简要介绍一下相关指令(大小写不敏感,但惯例为均大写),具体的还应参考官方文档:
FROM:指定基础镜像,必须是第一条指令。
MAINTAINER:指定镜像作者信息。
LABEL:指定镜像信息。
ENV:设置环境变量。
ADD & COPY:将本地文件复制进镜像,注意COPY只能是本地文件,ADD可以是url,并且自动解压缩。
RUN:运行指定的命令,每一个RUN指令都将在一个新的container里面运行,并提交为一个image作为下一个RUN的base,即层与层之间不会共用内存,所以如果要将多条命令联合起来执行则需加上&&。另外,在一个Dockerfile中可以包含多个RUN,按顺序执行。
CMD:在容器启动时要运行的命令。
首先根据自己常用操作系统pull官方系统文件,如1
2docker search -f stars=25 centos
docker pull centos
然后建立一个文件夹用于存放制作镜像过程中所用到的文件,下载软件的源码包并新建Dockerfile文件;
接下来制作镜像:1
docker build -t macs2:ubuntu.v1 .
其中:前面为镜像名称,后面为TAG,.表示当前目录,也可以使用全路径,目的就是找到Dockerfile文件。
由于需要网络环境,整体过程可能较慢。构建成功后,可docker images查看。
Docker Hub有大量现成镜像,随便搜索一个打开其Dockerfile察看,参考制作自己的镜像文件;
然后登陆docker login,成功后推送docker push wenlongshen/macs2:ubuntu.v1,至此,就可以在自己的docker hub里查看并管理了,同样地,也可以docker pull wenlongshen/macs2:ubuntu.v1到本地使用。
Singularity 使用
singularity有许多命令,常用的命令有,pull、run、exec、shell、build;
下载镜像1
singularity pull qiime2_core_2018.11.sif docker://qiime2/core:2018.11
载入数据1
singularity exec -B /share/exercise/qiime2/emp-single-end-sequences qiime2_core_2018.11.sif qiime tools import --type EMPSingleEndSequences --input-path /share/exercise/qiime2/emp-single-end-sequences --output-path emp-single-end-sequences.qza
因为在容器中是没有/share/exercise/qiime2/emp-single-end-sequences 这个绝对路径的,所以需要使用-B选项将宿主机的/share/exercise/qiime2/emp-single-end-sequences路径映射到容器中;
参考资料
- Docker —— 从入门到实践
- 生物信息分析流程
- singularity
- Singularity入门之安装
