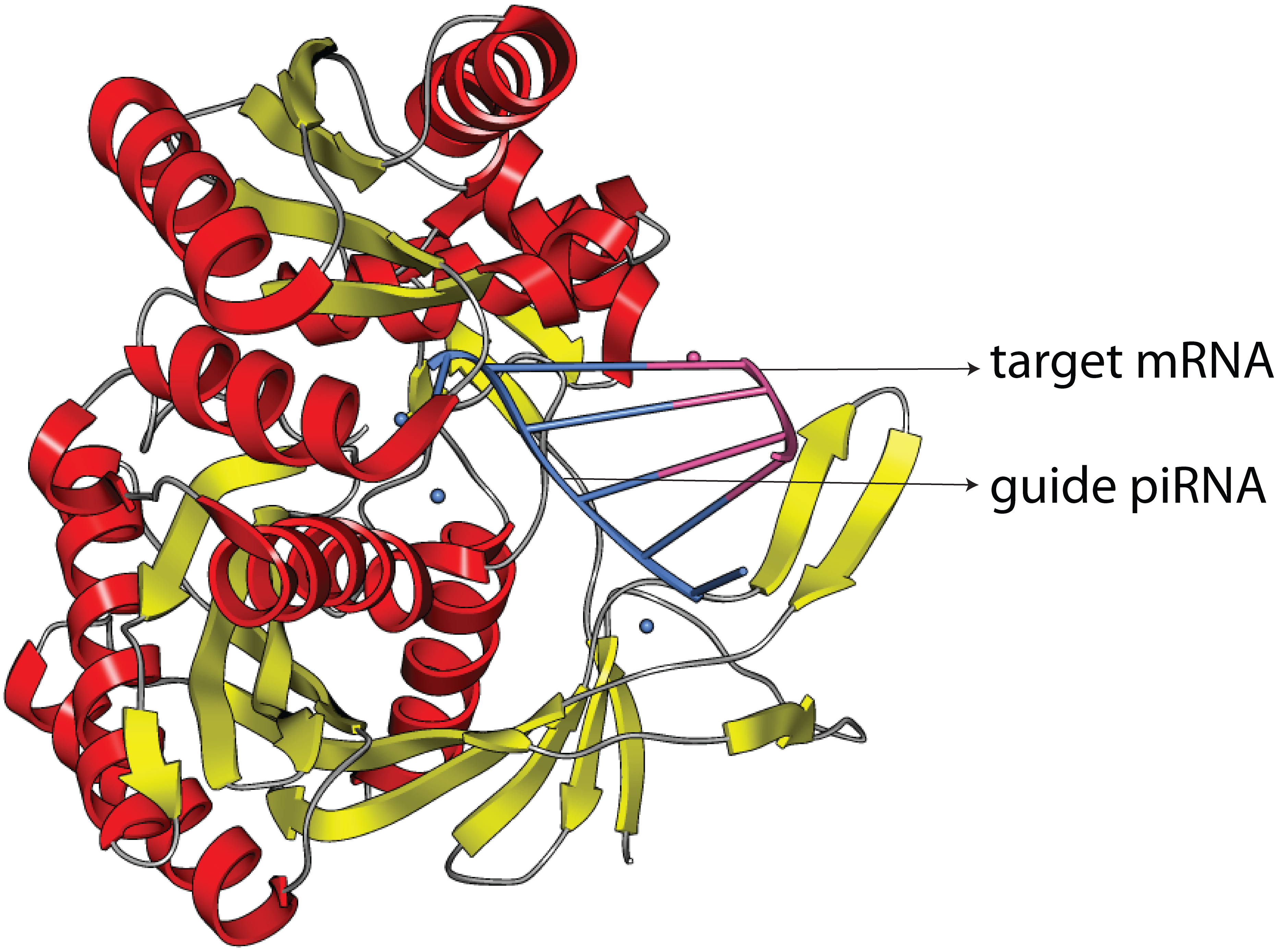
The spec file is an optional input to the runCA executive that launches the Celera Assembler pipeline. The spec files provides a convenient way to generate assemblies while documenting their parameters faithfully. The use of spec files is STRONGLY recommended.
Spec files参数解释
PBcR混合组装需要指定两个Spec配置文件: pacbio.spec(纠错)和asm.spec(组装)。这两个文件都包含特定的算法参数和计算机硬件参数,通常情况下算法参数可以忽略(此时将用软件默认值),但是计算机硬件参数需要根据实际情况调整。
所有参数均为option = value形式,其中的value为布尔型(boolean),即true=1,false=0。