CRISPR介绍和作用过程之前也学习总结过:CRISPR/Cas9。
Story
陆地棉基因组2015年完成测序,之后公布基因组信息,我所在实验也在从事CRISPR的工作,得益于技术革新和谢卡斌老师PNAS文章,CRISPR在棉花中的敲除首次在我们实验室获得成功,于是更多功能基因的CRISPR工作列入许多研究生的工作计划中,CRISPR编辑第一步就是目标基因的sgRNA设计。现有的绝大部分在线sgRNA设计主要是针对模式生物的,并不能用于非模式生物。但整个sgRNA设计原理是相通的也比较好理解:在目标基因序列的cds中匹配NGG的PAM区域,往前延伸20bp碱基就是一个理论的sgRNA位点,接下来所需考虑的就是这个sgRNA的特异性(不能编辑其他基因,20bp的碱基在几百M甚至G的基因组里面特异存在)和脱靶率(有时候为了保证特异性会允许一定数量的错配情况存在,这可能引起脱靶),所以后续会对这样的理论sgRNA位点在全基因组内比对,寻找可能的编辑位点。对于perl和python来说实现这样的功能并不是什么难事,华农动科学院谢老师用perl编写的可自行提供基因组的sgRNAcas9程序开发出来。
sgRNAcas9流程
sgRNAcas9软件功能介绍如下:
软件优点是可自行提供基因组文件,缺点是对酶切位点的计算较为麻烦。
偶然也发现软件作者收录了我早期一篇总结╰( ̄▽ ̄)╮
插播广告:该内容现有跟新,http://tiramisutes.github.io/2015/08/05/bio-online.html
软件下载
该软件是有perl程序编写,有windows和linux平台可供下载使用,根据相应平台自行下载:sgRNAcas9。
软件安装
修改可执行权限1
2
3chmod +x sgRNAcas9_3.0.5.pl
chmod +x -R Seqmap
chmod +x -R Usefull_Script
文件准备
基因组文件:genome.fa
基因组cds文件:genome_cds.fa
基因组注释文件:genome.gff3 (非必须)
将上述文件mv到sgRNAcas9软件所在目录,用绝对路径会报错。
代码运行
1 | perl sgRNAcas9_3.0.5.pl -i genome_cds.fa -x 20 -l 40 -m 60 -g genome.fa -o b -t s -v l -n 5 |
参数解释:
-i: 所需设计crispr敲除序列fa文件,可为多条序列;
-x: sgRNA长度,通常为20;
-l: GC含量下限;
-m: GC含量上限;GC含量一般为40%~60%。
-g: 基因组fa文件;
-o: 用DNA的哪条链作为crispr靶标位点搜寻,s正义链,a反义链,b双链;
-t: gRNA搜索模型,s单个gRNA,p一对gRNA搜寻;
-v: 操作系统类型,l为linux-64位,w为windows;
-n: 最大错配碱基数,一般为5。
-i参数设置全基因组cds的fa文件,-g参数设置去基因组fa文件就可得到上述提到的库文件。
结果解读
运行时间视所选物种基因组大小和目标基因多少相关;当基因组文件较大时windows下运行电脑易卡挂掉,最好在服务器下运行。
运行完后会生成report文件:sgRNAcas9.report_20.b.rhp.fa,包含以下内容: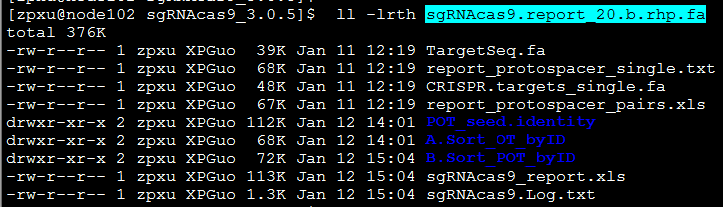
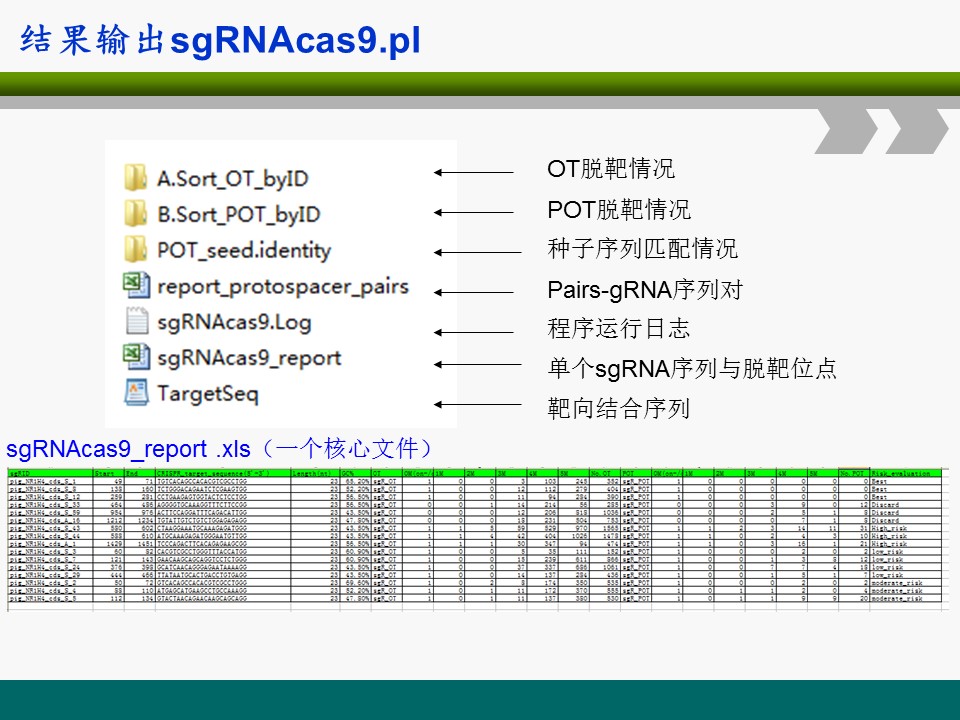
注:OT为靶标去除NGG后的脱靶情况,即NGG前面20个碱基的脱靶;POT为种子序列的脱靶情况,种子序列即NGG前面的12个碱基;
sgRNAcas9_report.xls文件里有个综合了GC含量,错配和特异性后的风险等级排序,可简单选取Best对应crispr靶标位点。
Discard > High_risk > moderate_risk > low_risk > repeat_sites_or_bad ? > Best
当0M(on-/off-)值为0时意味着脱靶,大于1则存在有靶标序列,其他数字表示所存在靶标数目;
脱靶位点注释
脱靶是要尽量避免的,但若存在风险还可根据基因组注释gff3文件对可能的脱靶位点进行注释。1
2perl ot2gtf.pl -i <input_OT> -g <gtf file> -o <output>
perl pot2gtf.pl -i <input_POT> -g <gtf file> -o <output>
CRISPR
CRISPR是华中农业大学生物信息学院陈玲玲老师团队开发的在线设计软件。输出结果可视化较好,包含有错配,靶标序列位置,酶切位点等信息。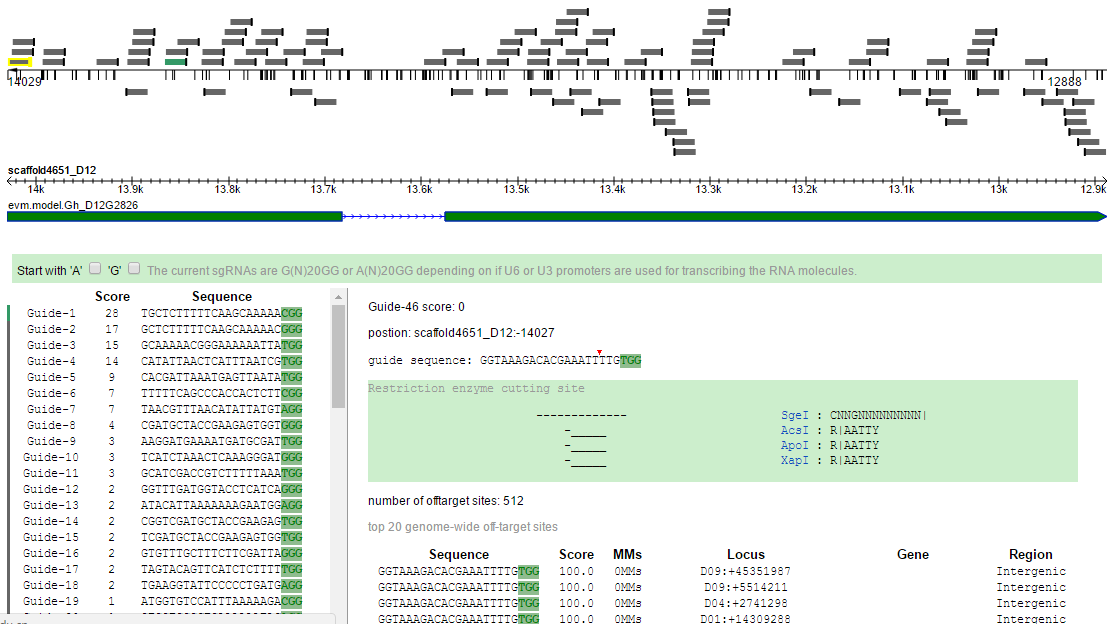
cas-designer
cas-designer也提供有在线版和命令行版,可自行提供基因组数据,官网有详细的安装教程,不再敖述。
比较坑的是其中一个组件Cas-OFFinder需要OpenCL-enabled device,而Centos6.0尽然不支持这种驱动,所以放弃。
总结
最终sgRNA位点可综合多个软件结果信息,筛选靠谱的结果;
